Oncogène - p53 Mutant exerce un effet dominant négatif en empêchant p53 de type sauvage de se lier à
Mutant p53 exerce un effet négatif dominant en empêchant p53 de type sauvage de se lier au promoteur de ses gènes cibles
Amy Willis 1. Eun Joo Jung 1. Therese Wakefield 1 et Xinbin Chen 1
1 Département de biologie cellulaire et UAB Comprehensive Cancer Center, l'Université d'Alabama à Birmingham, Birmingham, AL 35294-0005, États-Unis
p53, p53 mutante, l'arrêt du cycle cellulaire, la transcription
introduction
L'expression du mutant et p53 de type sauvage dans le double système inductible
Établissement de lignées cellulaires stables exprimant de manière inductible p53 mutant, p53 de type sauvage, ou les deux protéines simultanément. Utilisation du double système inductible, les lignées cellulaires ont été établies qui peuvent exprimer de manière inductible p53 de type sauvage et l'un des mutants p53 hot-spot: R175H (a), R248W (b), ou R273H (c). Les cellules ont été induites pour exprimer la protéine ni (piste 1), le mutant p53 seul (piste 2), p53 de type sauvage seul (piste 3), ou en même temps que les deux protéines (piste 4) pendant 24 h. Des extraits cellulaires ont été recueillies et des protéines résolues par SDS-PAGE. l'expression de p53 a été détectée par sondage avec des anticorps de p53 Pab1801 et PAb421. l'expression de p21 a été détectée avec l'anticorps polyclonal de p21. La membrane a été sondé avec l'actine comme un contrôle de chargement
pleine figure et la légende (113K)
Mutant et de type sauvage p53 interagissent physiquement et sont co-exprimées lorsque les deux sont exprimées dans le double système inductible
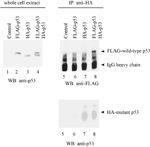
Mutant et p53 de type sauvage interagir physiquement in vivo. Des extraits cellulaires ont été préparés à partir de cellules (lignée cellulaire # 035; 208) non induit (pistes 1 et 5), induites pour exprimer le mutant p53 (R273H) HA marquée (pistes 3 et 7), p53 de type sauvage étiqueté FLAG-(pistes 2 et 6), ou les deux (pistes 4 et 8). Pour les voies 1-4, le Western Blot a été sondé avec un anticorps anti-p53. Pour les voies 5-8, les lysats cellulaires ont été immunoprécipités avec un anticorps anti-HA (pistes 5-8) et les protéines dans les immunoprécipités ont ensuite été détectée avec un anticorps anti-FLAG (panneau de droite sur le dessus) ou un anticorps anti-p53 (panneau de droite sur le fond)
pleine figure et la légende (81K)
Bien que l'expression de p53 mutant et de type sauvage a été sélectionné dans nos lignées cellulaires stables, nous voulions nous assurer que ces protéines ont été exprimées dans la même cellule. Cela a été réalisé en utilisant une coloration par immunofluorescence de mutant étiquetée HA p53 (R273H) et FLAG-étiqueté p53 de type sauvage. Comme représenté sur la figure 3. mutant p53 a été détectée par l'anticorps polyclonal anti-HA (rouge) et p53 de type sauvage par l'anticorps anti-FLAG monoclonal (vert) dans la même cellule lorsque les deux protéines ont été induites. Lorsque ces images ont été fusionnées, le signal fluorescent a été modifié, ce qui suggère que p53 mutant et de type sauvage ont été localisés dans le même compartiment de noyau. Par conséquent, la diminution de l'induction de p21 est probablement due à l'effet dominant négatif direct de p53 mutant sur p53 de type sauvage.
Mutant et p53 de type sauvage sont exprimés dans la même cellule. Les cellules ont été ensemencées dans des lames à chambre quatre puits, induite pendant 24 h dans un milieu d'induction appropriées et colorées pour mutant (anti-HA) et de type sauvage (anti-Flag) p53. tache nucléaire est bleu, tache mutant p53 est rouge, et tache p53 de type sauvage est vert
pleine figure et la légende (75K)
p53 Mutant inhibe la capacité de p53 de type sauvage à transactiver ses gènes cibles
pleine figure et la légende (130K)
Mutant p53 inhibe la capacité de p53 de type sauvage pour se lier à l'élément sensible à p53 dans ses gènes cibles

pleine figure et la légende (83K)
p53 Mutant réduit la capacité de p53 de type sauvage pour supprimer la prolifération des cellules
p53 Mutant inhibe la capacité de p53 de type sauvage pour supprimer la prolifération des cellules. Les cellules ont été amenées à exprimer ni protéines, mutant p53 seul, p53 de type sauvage seul, ou les deux en même temps. Les taux de croissance ont été évalués sur 5 jours
pleine figure et la légende (77K)
mutant dominant négatif p53 perturbe l'induction de l'arrêt du cycle cellulaire par p53 de type sauvage

p53 Mutant inhibe la capacité de p53 de type sauvage pour induire l'arrêt du cycle cellulaire. Les cellules ont été induites pour exprimer la protéine ni (témoin), p53 mutant seul, p53 de type sauvage seul, ou en même temps que les deux protéines (mutant + type sauvage). analyse de l'histogramme d'ADN a été utilisé pour déterminer le pourcentage de cellules dans chaque phase du cycle cellulaire tel que détecté par coloration à l'iodure de propidium d'ADN
pleine figure et la légende (183K)
Discussion
Pour déterminer le mécanisme de l'activité négative dominante, nous avons procédé à puce pour déterminer si p53 mutant empêche la protéine de type sauvage à partir spécifique de la séquence de liaison au promoteur de ses gènes cibles. Ce procédé est avantageux car il permet l'exploration de liaison à l'ADN in vivo. Le double système inductible permet la comparaison de cette liaison dans des cellules exprimant p53 de type sauvage seul à des cellules exprimant p53 de type sauvage en présence d'une p53 mutante. Ainsi, nos données ont fourni des preuves convaincantes que l'activité dominante négative de p53 mutante est l'inhibition de p53 de type sauvage pour se lier à l'élément sensible à p53 et par la suite, son activité de transcription.
matériaux et méthodes
Mise en place d'un système à double inductible et les lignées cellulaires stables
Immunoprécipitation et analyses occidentales
Les analyses de taux de croissance
Les cellules ont été ensemencées à raison d'environ 60 000 cellules / plaque de 60 mm. Les cellules ont été amenées à exprimer ni protéines, mutant p53 seul, p53 de type sauvage seul, ou les deux protéines simultanément. Le milieu a été remplacé tous les 72 h. Chaque jour pendant 5 jours, deux plaques pour chacune des quatre inductions ont été lavés avec DMEM pour éliminer les cellules mortes. Les cellules vivantes ont été trypsinisées et des échantillons de chaque plaque ont été comptés trois fois à l'aide d'un compteur de cellules Coulter (Coulter Corporation, Miami, FL, USA). Le nombre moyen de cellules provenant de chaque plaque a été calculée pour déterminer le taux de croissance pour chaque induction.
ADN analyse de l'histogramme
Les cellules ont été ensemencées à environ 200 000 cellules / plaque de 100 mm et induites pour exprimer ni protéine, p53 mutant seul, p53 de type sauvage seul, ou les deux protéines simultanément. Au bout de 3 jours après l'induction, les cellules flottantes dans les cellules de taille et en direct sur la plaque ont été recueillies et fixées avec de l'éthanol à 100% pendant au moins 1 h à 4 ° C. Les cellules fixées ont été centrifugées et remises en suspension à une concentration de 2 millions de cellules / ml dans du PBS avec 1 à 20 g / ml de RNase A et 50 g / ml d'iodure de propidium (Sigma Chemical Co. St Louis, MO, USA). Les cellules colorées ont été analysées dans un trieur de cellules activé par fluorescence (FACSCaliber; Becton Dickson, Menlo Park, CA, USA) dans les 4 h.
l'isolement de l'ARN et analyse Northern blot
Les cellules ont été ensemencées à raison d'environ 60% de confluence et induites pour exprimer ni protéine, p53 mutant seul, p53 de type sauvage seul, ou les deux protéines simultanément pendant 16 h. Les cellules ont été recueillies par raclage et les noyaux ont été extraits sur de la glace dans hypotonique tampon A (10 mM HEPES, 1,5 mM de MgCl2. KCl M 10 m, et un cocktail d'inhibiteurs de proteases) pendant 10 minutes, puis 0,5% de NP-40. L'ADN génomique et des protéines ont été réticulées avec 1% de formaldehyde pendant 10 minutes à température ambiante. Les échantillons ont été soumis à une sonication pour obtenir de 200 à 1000 pb fragments d'ADN. En tout, 1 / 50e de l'échantillon a été retiré pour l'entrée. Après clarification par centrifugation, les lysats soniqués ont été diluées avec un volume égal de 2 tampon RIPA (100 mM de Tris-HCl pH 7,4, 2% de NP-40, 0,5% de Na-désoxycholate, 300 m M de NaCl, 2 mM EDTA) et 1 . 100 protéase cocktail d'inhibiteurs et immunoprécipitées avec un anticorps anti-Flag M2, p53Pab 1801/421 Pab, ou Myc 9E10 comme témoin négatif. Les immunocomplexes ont été capturés par la protéine A / G billes de sépharose qui ont été saturés avec de l'ADN de sperme de saumon et lavé successivement avec du tampon TSE (0,1% de SDS, 1% de Triton X-100, 2 mM d'EDTA, 20 mM Tris-HCl pH 8,1) contenant du NaCl m 150 m, TSE contenant 500 mM de NaCl, le tampon III (0,25 m de LiCl, 1% de NP-40, 1% de désoxycholate de sodium, 1 mM d'EDTA, 10 mM Tris-HCl pH 8,1) et du tampon TE ( 10 m m Tris-HCl pH 8,0, 0,1 mM EDTA). Les immunocomplexes ont ensuite été éluées deux fois avec du tampon d'élution (SDS à 1%, 0,1 M NaHCO3) pendant 15 min à température ambiante. La réticulation a été inversée à 65 ° C pendant au moins 6 h. L'ADN a été purifié par extraction au phénol-chloroforme et précipitation à l'éthanol et remis en suspension dans 50 l de TE. PCR a été effectuée pour détecter le fragment de promoteur de p21 avec le p2151 d'amorces (de CAGGCTGTGGCTCTGATTGG) et 3' 5' p2131 d'amorces (de TCCAGAGTAACAGGCTAAGG). La PCR a été réalisée pour détecter l'ADN génomique MDM2 avec l'amorce 5' MDM2RE1F (GGGAGTTCAGGGTAAAGGTCA) et l'amorce 3' MDM2RE1R (CCTTTTACTGCAGTTTCG). PCR a été effectuée pour détecter le fragment de promoteur PIG3 avec l'amorce 5' PIG3 RE F (CCAACGGCTCCTTTCTCTTC) et l'amorce 3' PIG3 RE R (GCTTGACAGAAAGTGCGATTC).
immunofluorescence
Les références
Remerciements
Ce travail est soutenu en partie par des subventions du NIH 2 RO1 CA076069.