PUMAdb Valeur décomposition Aide Singulier
Documents d'aide connexes
- Aide référentiel. Explication du système de PUMAdb pour vous permettre d'enregistrer et de partager des fichiers à différentes étapes de l'analyse
- Formats de fichiers. Informations sur preclustering (.pcl), table de données en cluster (.cdt), arbre de gène (.gtr) et les fichiers arbre de tableau (.atr) généré dans le processus de données de regroupement
Vous pouvez utiliser SVD en cliquant sur le bouton « SVD » pour tout fichier .pcl dans votre référentiel. Il existe des documents d'aide fournis à la fois le dépôt et les formats de fichiers.
Pour utiliser SVD tel qu'il est mis en œuvre dans le cadre de la base de données, vous devrez d'abord mettre un fichier preclustering (de .pcl) dans votre référentiel. Bien que la SVD d'un ensemble de données est indépendante de l'ordre des gènes et des tableaux dans les données, un ordre significatif pourrait vous aider à corréler eigengenes dominantes avec des artefacts expérimentaux qui se superposent aux données ou avec des processus biologiques qui sont présentes dans les données. Pour cette raison, il est souvent avantageux de commander vos tableaux en utilisant une expérience ensemble ou une liste array (comme on pourrait déjà avoir fait pour une série chronologique) ou par des gènes en cluster et / ou des réseaux en utilisant le pipeline de regroupement de bases de données et la récupération des données en utilisant cet ordre.
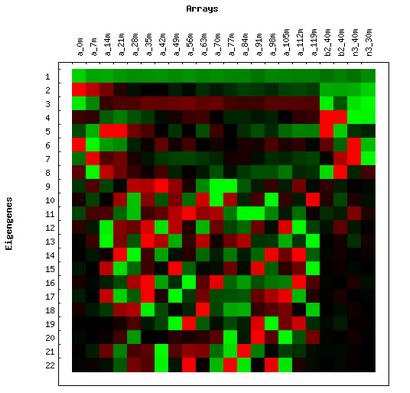
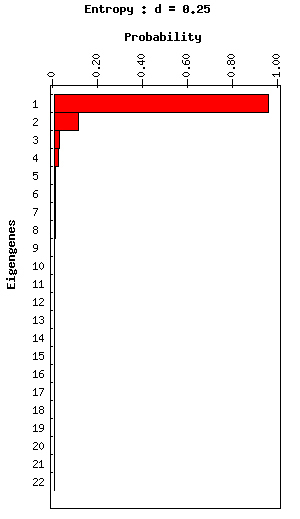
Figure 1. Affichage Raster de la Eigengenes (gauche) et Affichage de la barre graphique de la
Probabilités de Eigenexpression (droit) de levure Cycle cellulaire non centrée sur les données
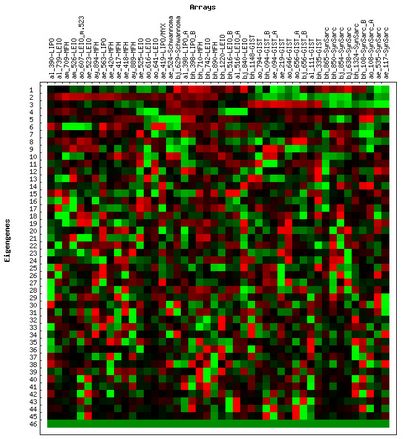
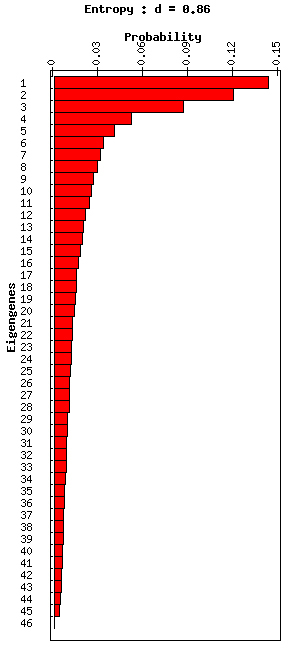
Figure 2. Affichage Raster de la Eigengenes (gauche) et Affichage de la barre graphique de la
Probabilités de Eigenexpression (droite) de Centré Sarcome Données humaines de tumeur
Une fois qu'un fichier .pcl a été préparé et enregistré, vous pouvez utiliser SVD en cliquant sur le bouton « SVD » dans votre référentiel.
Le logiciel affiche la matrice de eigengenes (la matrice la plus à gauche sur la figure 3) dans un affichage de trame rouge et verte le long d'un affichage graphique à barres de leurs probabilités correspondantes de eigenexpression.
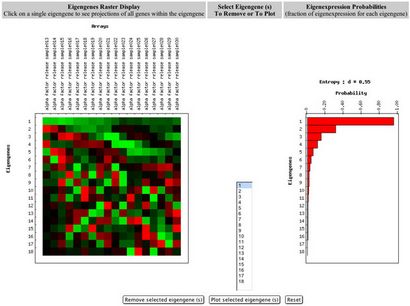
Figure 3. Outil de base de données pour la visualisation et l'utilisation de SVD dans l'analyse de données d'expression génique.
Chaque ligne de l'écran Raster Eigengenes représente un motif eigengene d'expression. La rangée supérieure dans la matrice de eigengenes est le premier eigengene, qui est celle qui contribue le plus à l'ensemble de données. A partir de cet écran, il y a plusieurs options qui sont expliquées ci-dessous:
- Terrain eigengene choisi (s)
- Voir la projection de l'une quelconque des gènes eigengene
- Retirer eigengene sélectionné (s)
Chaque ligne du graphique à barres (du côté droit de la figure 3) représente la probabilité de eigenexpression du eigengene correspondant (et eigenarray). Par exemple, la première barre (supérieure plus) dans le tableau est la probabilité de eigenexpression du premier eigengene (et aussi le premier eigenarray). Il y a plus d'informations sur la probabilité de eigenexpression et de l'entropie plus loin dans ce document.
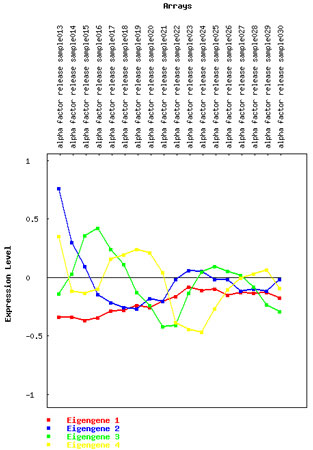
Figure 4. Tracer montrant le comportement de quatre eigengenes.
Cliquez sur une des eigengenes (lignes) dans l'affichage de la trame de la matrice de eigengenes pour afficher les projections de tous les gènes triées de votre ensemble de données avec ce eigengene. Cet écran vous permet de choisir les deux sous-ensembles de gènes de votre ensemble de données, avec la plus grande projection positive ou négative sur le eigengene sélectionné afin que vous puissiez les analyser plus loin. Cet affichage est illustré à la figure 5.
Utilisez la section « Sélection des groupes de données » pour sélectionner les données que vous pouvez entrer dans votre dépôt en tant que fichier .pcl ou simplement télécharger sur votre ordinateur de bureau. Ne pas oublier que vous pouvez sélectionner des gènes qui ont des projections élevées dans les deux directions positives et négatives. Vous pouvez ensuite regrouper ou encore analyser les données pour les gènes à rechercher des modèles. Il y a plus d'informations sur les projections de gènes plus loin dans ce document.
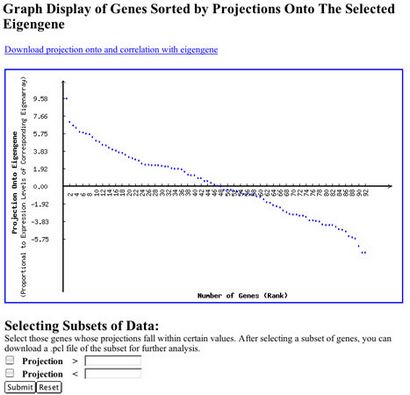
Figure 5. Projection de gènes au sein d'un eigengene. Cette image montre comment tous les gènes dans un ensemble de données sont projetées sur un eigengene donné. Ceci est une façon de déterminer les gènes dont l'expression est significativement contribué par un eigengene.
La suppression d'un eigengene est mathemetically équivalente à régler le niveau de eigenexpression de cette eigengene (et en même temps, son correspondant eigenarray) à zéro. Les trois matrices (la matrice de eigenarrays, la matrice et la matrice eigenexpression eigengene) sont alors mulitplied à nouveau pour reconstruire un ensemble de données de puces à ADN avec les effets de la eigengene (et son eigenarray correspondant) enlevé.
Après la suppression d'un eigengene, les gènes et les tableaux qui étaient dans l'ensemble de données de puces à ADN d'origine sont toujours là, mais les données d'expression elle-même a changé. Par exemple, un gène qui a la majeure partie de sa contribution d'une eigengene filtrée semble avoir un zéro expression presque constante sur tous les tableaux. Étant donné que les valeurs de données sont maintenant changées, si des filtres basés sur des valeurs de données sont importantes pour votre analyse, vous voudrez peut-être filtrer à nouveau vos données reconstruites.
Figure 6. Schéma (et mathématiquement inexact!) Représentation de la façon dont SVD trouve la « vue » des données qui capture la plus grande variance.
Ces artefacts peuvent ensuite être retirés de l'ensemble des données d'expression (sans écarter les données d'un quelconque des gènes ou des tableaux) en filtrant le eigengene correspondant (s) et eigenarray (s). Cette normalisation de données SVD, où les additifs et éventuellement des artefacts expérimentaux multiplicative sont détectés et filtrés, permet une meilleure analyse plus poussée par des méthodes telles que la classification hiérarchique, qui sont sensibles à la présence d'un artefact qui est superposé sur les données.
Deux exemples sont présentés ci-dessous:
La matrice d'expression de données sous forme de tableau l'expression des deux gènes et les tableaux: Les lignes de la matrice d'expression de données ventilent l'expression de chaque gène dans l'ensemble de données dans tous les tableaux, les colonnes et # 8211 l'expression de chaque tableau dans tous les gènes.
Les niveaux Eigenexpression
La matrice W a des dimensions N xN et est appelé la matrice des niveaux Eigenexpression. Chaque entrée enregistre l'expression d'un eigengene dans un eigenarray. En d'autres termes, il fournit un coefficient pour le rôle d'un eigengene joue dans un eigenarray (et vice-versa). La première entrée dans la matrice des niveaux de eigenexpression enregistre le niveau d'expression de la première eigengene dans la première eigenarray.
Chaque niveau eigenexpression représente la dominance du eigengene et eigenarray correspondant: le plus élevé est le niveau de eigenexpression, le plus dominant sont ces eigengene et eigenarray dans les données. Cela signifie, par exemple, que le premier eigengene et eigenarray sont toujours plus dominante que ou comme également dominant en tant que deuxième eigengene et eigenarray.
Les probabilités de eigenexpression qui sont présentés dans ce graphique à barres sont calculées à partir des niveaux de eigenexpression qui apparaissent dans la matrice diagonale du SVD de l'ensemble de données (voir la figure 8). La probabilité de eigenexpression indique l'importance d'un eigengene et son correspondant eigenarray en termes de la fraction de l'ensemble des informations d'expression qu'ils capturent dans l'ensemble de données. Il peut être considéré comme la probabilité que ce modèle eigengene se manifeste en tant que composant de l'expression de l'une quelconque des gènes. Dans le même temps, il peut également être considéré comme étant la probabilité que le motif eigenarray correspondant se manifeste en tant que composant de l'expression de l'une quelconque des tableaux. Les probabilités sont calculées en divisant le carré de la L-ième valeur propre (à partir de la matrice W) par la somme des carrés de toutes les valeurs propres et ils sont présentés sous forme de graphique à barres rouge à droite sur la figure 1.
Software présente la « entropie » de l'ensemble de données dans la légende du graphique à barres (par exemple, voir les figures 1-3 ci-dessus). L'entropie de l'ensemble de données mesure la complexité des données à partir de la distribution de l'expression globale entre les différents eigengenes et eigenarrays correspondant, et est calculée à partir des probabilités de eigenexpression de l'ensemble de données. L'entropie d'un ensemble de données ordonné et redondant, dans lequel toute expression est capturée par un seul eigengene et son eigenarray correspondant, est égal à 1. L'entropie d'un ensemble de données désordonnée et aléatoire, où sont également exprimés tous eigengenes et eigenarrays, est égal à 0. Généralement, l'entropie d'un ensemble de données d'expression non centré est d'environ 0,1 à 0,3, et l'entropie d'un ensemble de données d'expression centrée est d'environ 0,75 à 0,95. Filtrage eigengene et modèles eigenarray sur l'ensemble de données changeront l'entropie de l'ensemble de données.
Le dernier des trois matrices générées par SVD est la matrice U. Il y a M rangées contenant des données pour des gènes dans l'ensemble de données d'origine (la première rangée correspond au premier gène dans l'ensemble de données, la deuxième rangée correspond à la deuxième gène dans l'ensemble de données, etc.). Chaque colonne représente un groupement eigen (la première colonne correspondant à la première eigengene, la deuxième colonne correspond à la seconde eigengene, etc.). Chaque cellule de la matrice donne le coefficient par lequel le produit de W x V T doit être multiplié pour obtenir la quantité que l'eigengene contribue au vecteur de données pour ce gène. Ce qui est vraiment juste le long chemin de réaffirmer que notre ensemble de données d'origine peut être représentée comme suit:
Qu'est-ce que Superposition d'expression des données signifie?
Dans le cadre de SVD, l'expression de chaque gène et matrice est une superposition (ou une somme pondérée) de l'ensemble des eigengenes et eigenarrays, respectivement. On pourrait penser à la séparation mathématique SVD des profils d'expression des gènes et des tableaux dans eigengenes et eigenarrays correspondantes, respectivement, en tant que tentative de percer le signal global d'expression dans ses composants de génération: Les procédés expérimentaux et biologiques indépendants, et les états cellulaires correspondants. En d'autres termes, SVD peut être utilisé pour tenter de décrire les données d'expression comme le résultat d'un réseau simple, où quelques sources indépendantes d'expression, expérimentales ou biologiques, affectent tous les gènes dans l'ensemble de données (figure 10).
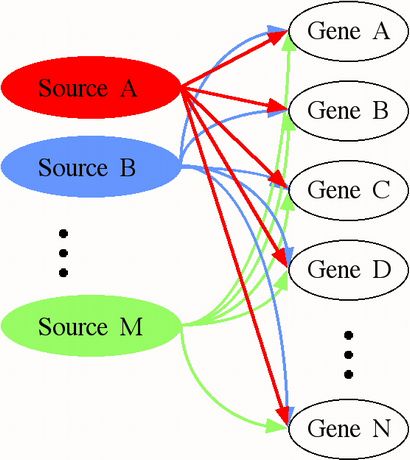
Figure 10. SVD peut être utilisé pour tenter de décrire les données d'expression comme le résultat d'un réseau de processus. Chaque « source » représente un processus (soit biologique ou artifactual) qui a un effet sur l'expression de chaque gène. L'effet de tout processus peut être grand ou petit, positif ou négatif.
Les projections de gènes sur et corrélations avec Eigengene sélectionnés.
Notez que la projection d'un gène sur un eigengene par rapport à celle d'un autre gène est listé dans le eigenarray qui correspond à cette eigengene. Cela signifie que la projection de tous les gènes dans l'ensemble de données sur un eigengene sont linéairement proportionnelle à la eigenarray correspondant.
La similitude de la configuration de l'expression du gène à la configuration de l'expression de l'eigengene est mesurée par « corrélation de gène avec le eigengene » (figure 11). Vous pouvez penser à cette corrélation en termes géométriques comme le cosinus de l'angle entre le gène et la eigengene, chacun représentant un vecteur dans l'espace. Encore une fois, vous voulez savoir s'il existe un thème biologique cohérent qui se reflète dans les annotations des gènes, les plus grandes corrélations positives avec le eigengene (avec les modèles les plus semblables à celle de la eigengene). Et vous voulez aussi voir s'il existe un thème biologique cohérente distincte reflétée dans les annotations des gènes avec les plus grandes corrélations négatives (avec les modèles les plus semblables aux modèles qui est antiparallèle à celle de la eigengene).
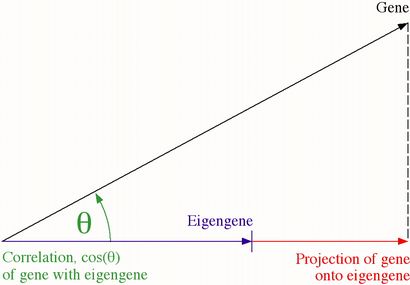
Figure 11. Description Géométrique la projection d'un gène sur et de corrélation avec un Eigengene
Pour ceux qui ne l'ont pas dû multiplier les matrices depuis le lycée, voici un très bref de recyclage qui pourraient vous aider à comprendre comment votre ensemble de données d'origine concerne les trois matrices générées par SVD. Quand on multiplie les deux matrices ci-dessous, nous obtenons un 2 par 2 matrice comme suit:
Nous pouvons faire le même type d'opération avec des nombres, comme illustré ci-dessous:
Quand on multiplie les trois matrices ci-dessous, nous obtenons un 3 par 2 matrice comme suit:
Et avec des chiffres: